近日,系统分类学领域著名国际刊物《Systematic Biology》在线发表了北京师范大学生态系核桃研究团队关于幽灵基因渐渗对物种树推断和分化时间估计影响的论文。
近年来,随着基因组测序数据越来越普遍,人们发现不同物种之间的基因流动(也称为基因渐渗)广泛存在于生命之树的各个枝杈。然而人们在利用分子序列信息构建物种树时,经常只考虑溯祖随机性的影响而忽略了种间基因流的作用。或者说,研究者普遍认为,一定程度的基因流对系统发育重建不会产生实质性的影响,可以安全地被忽略。但这个前提假设是否成立却很少受到人们的关注。此外,在系统发育重建工作中,被研究的物种通常只占目前存在或已经灭绝的所有物种的一小部分。因此,广泛存在于生命之树的基因渐渗很可能涉及“幽灵”物种,即未采样、未知或已灭绝的谱系。然而,幽灵基因渐渗对物种树估计的影响更是很少被研究。在本文,作者使用数学分析和模拟探讨了基于多物种溯祖模型的物种树方法对基因渐渗(幽灵和/或现存谱系间)的稳健性。
作者发现许多关于现存物种间基因渐渗的结果可以很容易地扩展到幽灵基因渐渗的情形,例如不完全谱系分选和基因渐渗对异常基因树(AGT)发生的可能性具有强交互作用:当不完全谱系分选的风险很高时,即使微弱的种间基因流也会导致错误的物种树;而当不完全谱系分选潜在影响很弱时,即使非常高水平的基因渗入(例如渐渗比例大于50%)也不一定会使得物种树拓扑关系产生偏差(参见图1)。这个结果意味着高水平的不完全谱系分选不仅增加了当前流行的系统发育推断方法准确估计物种树拓扑结构的难度,而且强化了基因渐渗(包括幽灵渐渗)误导种间相互关系推测的可能性。
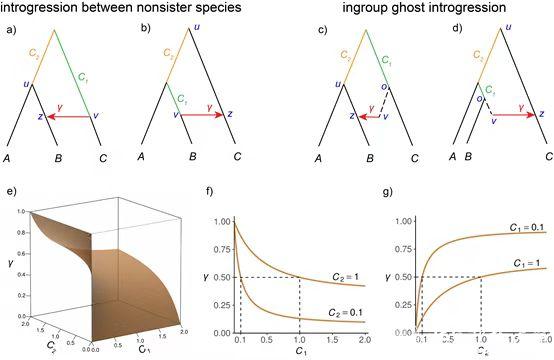
图1 非姐妹种间基因渐渗以及与之相对应的幽灵渐渗情景导致的异常带。a-b)代表不同渐渗方向(C->B,内向;B->C,外向)的非姐妹种间基因流情景。c-d)代表分别与a、b情景相似的由内群幽灵支系(虚线代表的ov边)介导的基因渐渗情景。对于所有的四个情景,考虑三个参数(γ,C1,C2),其中Ci为溯祖单位的枝长,为渐渗比例。C2代表物种树内枝的长度,而C1则在不同情景中含义不同。e)根据基因树拓扑概率划分参数空间,黄色曲面上方的空间,即P(G1=((a,b),c))<P(G2=((b,c)a)),代表异常带。固定C2(f)或C1(g)后,图e参数空间的二维截面。黄色曲线代表使得P(G1)=P(G2) 的“转折点”。
简易物种树法(ASTRAL)和全似然法(*BEAST)的相对表现在不同的基因渐渗情景下有所不同,后者在某些幽灵渐渗情形下表现更好,而前者对非姐妹种之间的基因流更稳健。当一个外群幽灵(定义为在被调查的最基础物种之前分化的谱系)作为渐渗基因的供体时,被调查物种之间的总体分化时间通常会被高估,而内群渐渗只会导致物种分化时间被低估。在许多内群渐渗(可能涉及幽灵谱系)的情景下,不完全谱系分选越强,估计总分化时间的准确性就越高,尽管物种树的拓扑结构更容易受到基因渐渗的影响
基因渐渗的方向对异常基因树(AGT)的发生率有显著影响,因为它不仅决定了在幽灵基因渗入下异常区是否可以存在,而且还决定了在非姐妹物种之间发生基因渐渗时异常区的大小。种间基因流方向对于物种分化时间的估计同样也很重要。当非姐妹物种之间的渐渗方向(如图2所示)是从外群到内群谱系时,随着渐渗强度的增加,物种分化时间的估计值显示出先减小然后再增加到真实值的趋势。而当基因渐渗方向是外向时,物种分化时间估计随着基因渐渗程度的增加而单调减少到姐妹物种的分化时间。
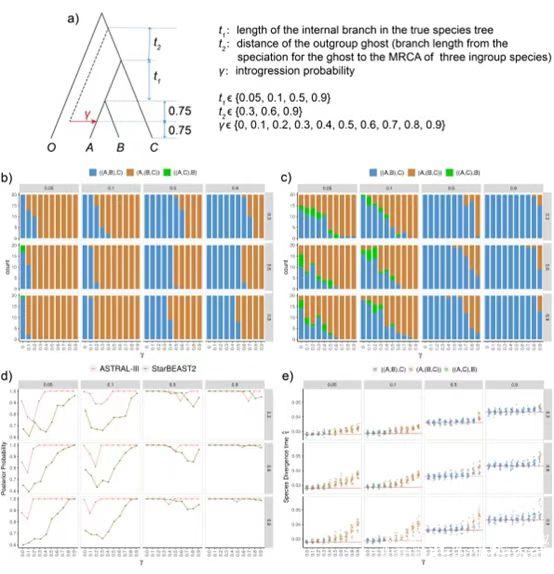
图2 渐渗方向为外群到内群(C->B)的非姐妹种间基因渐渗以及相应的幽灵渐渗情景的模拟结果。a)模拟情景和参数设置;b-e)模拟结果。图片顶部和右侧的分面标签分别代表t1和t2,x轴代表γ,每组参数组合设置20次重复。两种情景在相同参数设置下会产生相同的基因树分布,因而共享物种树推断结果。b)ASTRAL-III推断的物种树拓扑。c)StarBEAST2推断的物种树拓扑。d)两种方法推断的物种树支持率,每个点代表20次重复的平均值。e)A、B和C物种最近共同祖先的分化时间估计。点和红线分别代表估计值和真实值。
该研究于2022年7月7日正式发表在中科院一区刊物《Systematic Biology》上(2022年最新影响因子9.16,5年影响因子14.478)。Systematic Biology创刊于1952年,是系统生物学家学会的官方期刊,每年发文量只有80篇左右。北京师范大学生态系博士研究生庞晓旭为该论文的第一作者和共同通讯作者,张大勇教授为共同通讯作者。该研究得到了国家重点研发计划项目(2017YFA0605104)、国家自然科学基金项目(31421063)、“111”高校学科创新引智计划项目(B13008)以及北京市“地表过程”高精尖项目的支持。
文章链接:https://academic.oup.com/sysbio/advance-article/doi/10.1093/sysbio/syac047/6633313